Après quelques essais et erreurs, j'ai découvert comment interpréter les résultats de scipy.signal.deconvolve() et je poste mes découvertes en guise de réponse.
Commençons par un exemple de code fonctionnel
import numpy as np
import scipy.signal
import matplotlib.pyplot as plt
# let the signal be box-like
signal = np.repeat([0., 1., 0.], 100)
# and use a gaussian filter
# the filter should be shorter than the signal
# the filter should be such that it's much bigger then zero everywhere
gauss = np.exp(-( (np.linspace(0,50)-25.)/float(12))**2 )
print gauss.min() # = 0.013 >> 0
# calculate the convolution (np.convolve and scipy.signal.convolve identical)
# the keywordargument mode="same" ensures that the convolution spans the same
# shape as the input array.
#filtered = scipy.signal.convolve(signal, gauss, mode='same')
filtered = np.convolve(signal, gauss, mode='same')
deconv, _ = scipy.signal.deconvolve( filtered, gauss )
#the deconvolution has n = len(signal) - len(gauss) + 1 points
n = len(signal)-len(gauss)+1
# so we need to expand it by
s = (len(signal)-n)/2
#on both sides.
deconv_res = np.zeros(len(signal))
deconv_res[s:len(signal)-s-1] = deconv
deconv = deconv_res
# now deconv contains the deconvolution
# expanded to the original shape (filled with zeros)
#### Plot ####
fig , ax = plt.subplots(nrows=4, figsize=(6,7))
ax[0].plot(signal, color="#907700", label="original", lw=3 )
ax[1].plot(gauss, color="#68934e", label="gauss filter", lw=3 )
# we need to divide by the sum of the filter window to get the convolution normalized to 1
ax[2].plot(filtered/np.sum(gauss), color="#325cab", label="convoluted" , lw=3 )
ax[3].plot(deconv, color="#ab4232", label="deconvoluted", lw=3 )
for i in range(len(ax)):
ax[i].set_xlim([0, len(signal)])
ax[i].set_ylim([-0.07, 1.2])
ax[i].legend(loc=1, fontsize=11)
if i != len(ax)-1 :
ax[i].set_xticklabels([])
plt.savefig(__file__ + ".png")
plt.show()
Ce code produit l'image suivante, montrant exactement ce que nous voulons (Deconvolve(Convolve(signal,gauss) , gauss) == signal )
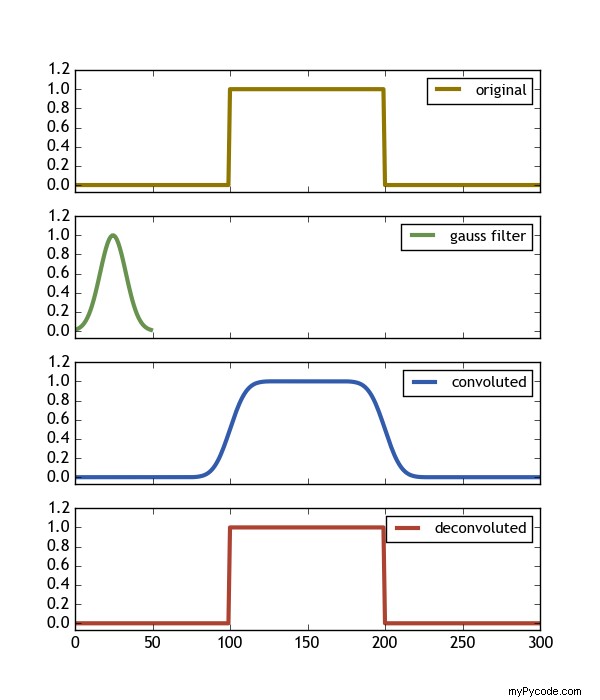
Voici quelques résultats importants :
- Le filtre doit être plus court que le signal
- Le filtre doit être beaucoup plus grand que zéro partout (ici> 0,013 est suffisant)
- Utilisation de l'argument de mot-clé
mode = 'same'à la convolution garantit qu'elle vit sur la même forme de tableau que le signal. - La déconvolution a
n = len(signal) - len(gauss) + 1points.Donc, afin de le laisser également résider sur la même forme de tableau d'origine, nous devons l'étendre des = (len(signal)-n)/2des deux côtés.
Bien sûr, d'autres découvertes, commentaires et suggestions à cette question sont toujours les bienvenus.
Comme écrit dans les commentaires, je ne peux pas aider avec l'exemple que vous avez posté à l'origine. Comme @Stelios l'a souligné, la déconvolution pourrait ne pas fonctionner en raison de problèmes numériques.
Je peux cependant reproduire l'exemple que vous avez posté dans votre Edit :
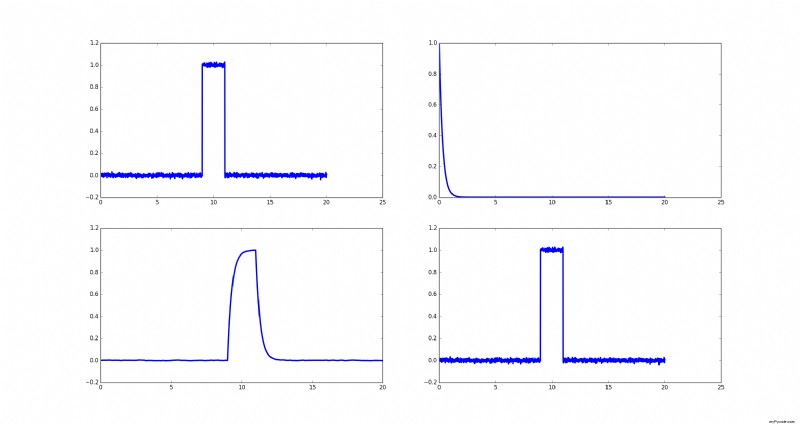
C'est le code qui est une traduction directe du code source matlab :
import numpy as np
import scipy.signal
import matplotlib.pyplot as plt
x = np.arange(0., 20.01, 0.01)
y = np.zeros(len(x))
y[900:1100] = 1.
y += 0.01 * np.random.randn(len(y))
c = np.exp(-(np.arange(len(y))) / 30.)
yc = scipy.signal.convolve(y, c, mode='full') / c.sum()
ydc, remainder = scipy.signal.deconvolve(yc, c)
ydc *= c.sum()
fig, ax = plt.subplots(nrows=2, ncols=2, figsize=(4, 4))
ax[0][0].plot(x, y, label="original y", lw=3)
ax[0][1].plot(x, c, label="c", lw=3)
ax[1][0].plot(x[0:2000], yc[0:2000], label="yc", lw=3)
ax[1][1].plot(x, ydc, label="recovered y", lw=3)
plt.show()