Questo, penso possa funzionare come punto di partenza. Non sono un esperto di elaborazione del segnale, ma l'ho provato su un segnale generato Y sembra abbastanza simile al tuo e uno con molto più rumore:
from scipy.signal import convolve
import numpy as np
from matplotlib import pyplot as plt
#Obtaining derivative
kernel = [1, 0, -1]
dY = convolve(Y, kernel, 'valid')
#Checking for sign-flipping
S = np.sign(dY)
ddS = convolve(S, kernel, 'valid')
#These candidates are basically all negative slope positions
#Add one since using 'valid' shrinks the arrays
candidates = np.where(dY < 0)[0] + (len(kernel) - 1)
#Here they are filtered on actually being the final such position in a run of
#negative slopes
peaks = sorted(set(candidates).intersection(np.where(ddS == 2)[0] + 1))
plt.plot(Y)
#If you need a simple filter on peak size you could use:
alpha = -0.0025
peaks = np.array(peaks)[Y[peaks] < alpha]
plt.scatter(peaks, Y[peaks], marker='x', color='g', s=40)
I risultati del campione: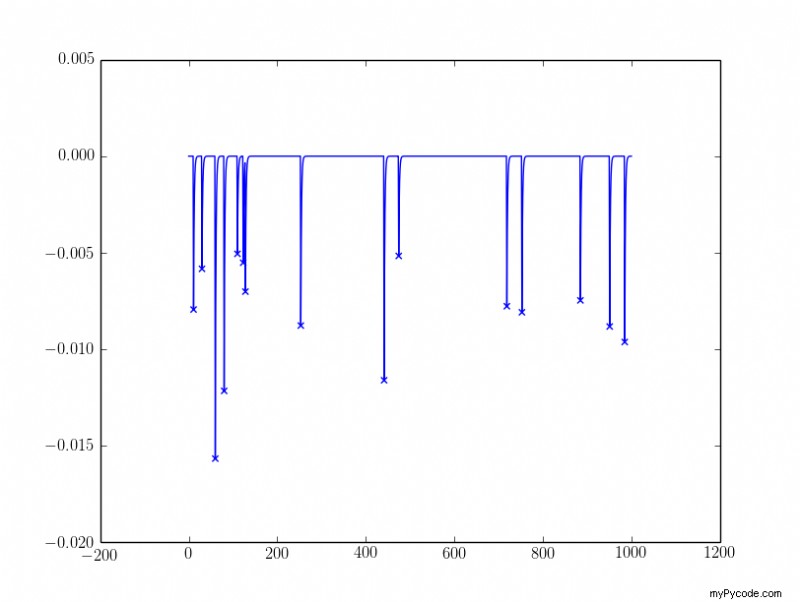 Per quello rumoroso ho filtrato i picchi con
Per quello rumoroso ho filtrato i picchi con alpha :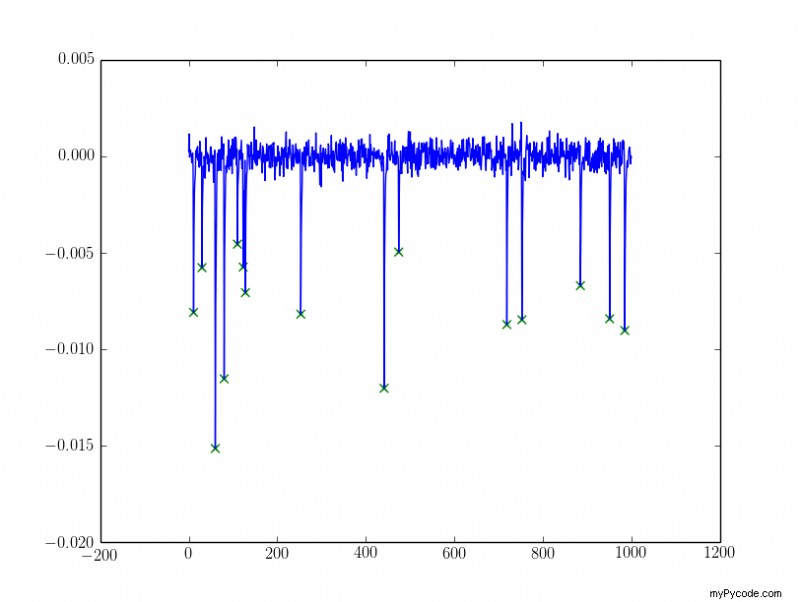
Se il alpha ha bisogno di più sofisticatezza potresti provare a impostare dinamicamente l'alfa dai picchi scoperti usando ad es. ipotesi sul fatto che siano una gaussiana mista (la mia preferita è la soglia di Otsu, esiste in cv e skimage ) o una sorta di clustering (k-means potrebbe funzionare).
E per riferimento, questo l'ho usato per generare il segnale:
Y = np.zeros(1000)
def peaker(Y, alpha=0.01, df=2, loc=-0.005, size=-.0015, threshold=0.001, decay=0.5):
peaking = False
for i, v in enumerate(Y):
if not peaking:
peaking = np.random.random() < alpha
if peaking:
Y[i] = loc + size * np.random.chisquare(df=2)
continue
elif Y[i - 1] < threshold:
peaking = False
if i > 0:
Y[i] = Y[i - 1] * decay
peaker(Y)
EDIT:supporto per il degrado della linea di base
Ho simulato una linea di base inclinata in questo modo:
Z = np.log2(np.arange(Y.size) + 100) * 0.001
Y = Y + Z[::-1] - Z[-1]
Quindi per rilevare con un'alfa fissa (nota che ho cambiato segno sull'alfa ):
from scipy.signal import medfilt
alpha = 0.0025
Ybase = medfilt(Y, 51) # 51 should be large in comparison to your peak X-axis lengths and an odd number.
peaks = np.array(peaks)[Ybase[peaks] - Y[peaks] > alpha]
Con il risultato seguente (la linea di base viene tracciata come linea nera tratteggiata):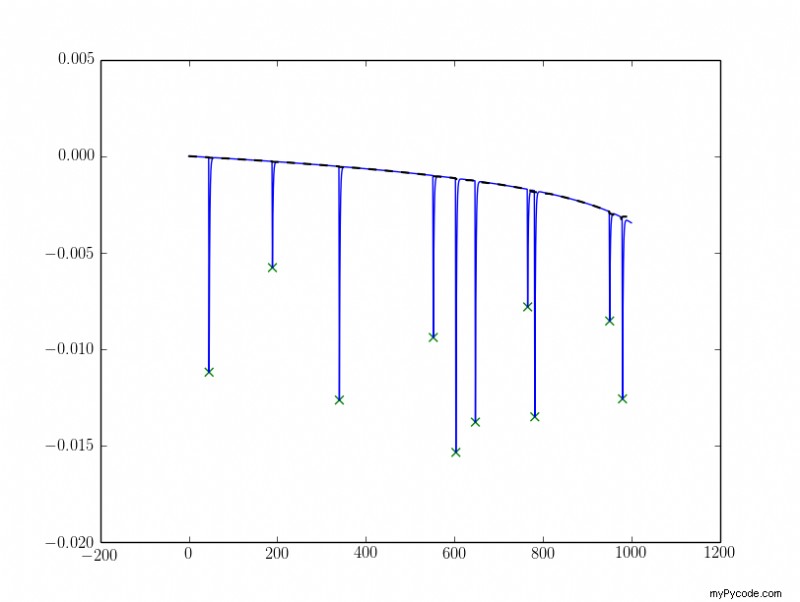
EDIT 2:Semplificazione e un commento
Ho semplificato il codice per utilizzare un kernel per entrambi convolve come ha commentato @skymandr. Questo ha anche rimosso il numero magico nella regolazione del restringimento in modo che qualsiasi dimensione del kernel dovrebbe fare.
Per la scelta di "valid" come opzione per convolve . Probabilmente avrebbe funzionato altrettanto bene con "same" , ma scelgo "valid" quindi non ho dovuto pensare alle condizioni del bordo e se l'algoritmo fosse in grado di rilevare picchi spurio lì.
A partire dalla versione 1.1 di SciPy, puoi anche usare find_peaks:
import numpy as np
import matplotlib.pyplot as plt
from scipy.signal import find_peaks
np.random.seed(0)
Y = np.zeros(1000)
# insert @deinonychusaur's peaker function here
peaker(Y)
# make data noisy
Y = Y + 10e-4 * np.random.randn(len(Y))
# find_peaks gets the maxima, so we multiply our signal by -1
Y *= -1
# get the actual peaks
peaks, _ = find_peaks(Y, height=0.002)
# multiply back for plotting purposes
Y *= -1
plt.plot(Y)
plt.plot(peaks, Y[peaks], "x")
plt.show()
Questo verrà tracciato (nota che usiamo height=0.002 che troverà solo picchi superiori a 0,002):
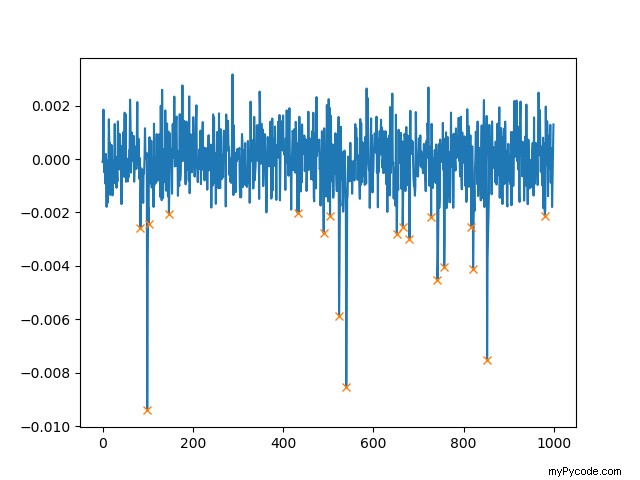
Oltre a height , possiamo anche impostare la distanza minima tra due picchi. Se usi distance=100 , la trama appare quindi come segue:
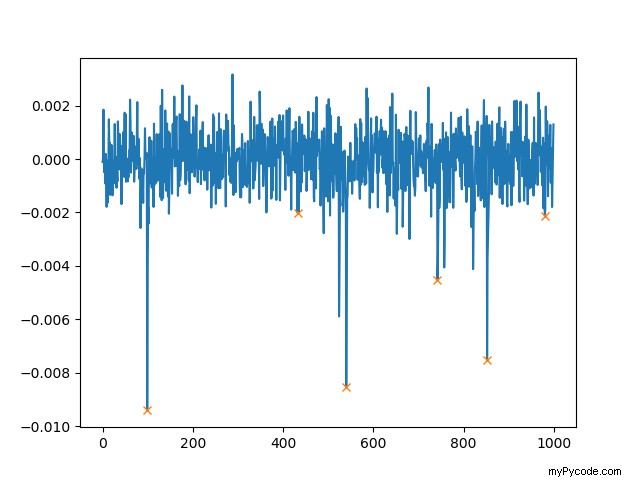
Puoi usare
peaks, _ = find_peaks(Y, height=0.002, distance=100)
nel codice sopra.